
- Forfatter Miles Stephen stephen@answers-science.com.
- Public 2023-12-15 23:39.
- Sist endret 2025-06-01 05:05.
DNA-konsentrasjon er estimert av måle absorbansen ved 260nm, justere A260 måling for turbiditet ( målt etter absorbans ved 320nm), multiplisere av fortynningsfaktoren, og ved hjelp av forholdet som en A260 på 1,0 = 50 ug/ml rent dsDNA.
Folk spør også, hvordan finner du konsentrasjonen og renheten til DNA?
Å evaluere DNA-renhet , mål absorbansen fra 230nm til 320nm for å oppdage andre mulige forurensninger. Den vanligste renhetsberegning er forholdet mellom absorbansen ved 260 nm delt på avlesningen ved 280 nm. God kvalitet DNA vil ha en A260/EN280 forhold på 1,7-2,0.
På samme måte, hva er en god DNA-konsentrasjon? EN god kvalitet DNA prøven skal ha en A260/EN280 forhold på 1,7-2,0 og en A260/EN230 forhold på over 1,5, men siden følsomheten til forskjellige teknikker for disse forurensningene varierer, bør disse verdiene kun tas som en veiledning for renheten til prøven.
Angående dette, hvordan beregner NanoDrop DNA-konsentrasjon?
Du multipliserer verdien av absorbans ved 260 nm med en fast faktor (det er 50 for DNA ) og du får DNA-konsentrasjon . Voilá. (Ok, teknisk sett er det A260 til en 10 mm tykk prøve som multipliseres med 50; NanoDrop uttrykker A260 som om prøven var 10 mm tykk, som i en standard kyvette).
Hvorfor måtte vi bruke et spektrofotometer for å kvantifisere vårt DNA?
EN spektrofotometer er i stand til å bestemme de gjennomsnittlige konsentrasjonene av nukleinsyrene DNA eller RNA tilstede i en blanding, så vel som deres renhet. Ved hjelp av Beer-Lambert-loven det er mulig å relatere mengden lys som absorberes til konsentrasjonen av det absorberende molekylet.
Anbefalt:
Hvordan måler du diameteren til en sylinder ved hjelp av en vernier caliper?
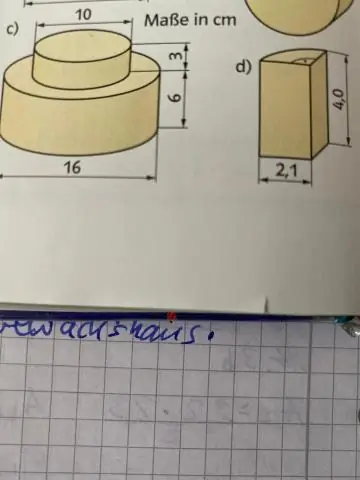
For å finne lengden på sylinderen/objektet: Hold sylinderen fra endene ved hjelp av de nedre kjevene på vernier-kaliperen. Legg merke til avlesningen på hovedskalaen som ligger like til venstre for nullmerket for nollskalaen. Se nå etter merket på vernier-skalaen som er på linje med et merke på hovedskalaen
Hvordan finner du radiusen til en sirkel ved hjelp av pi?
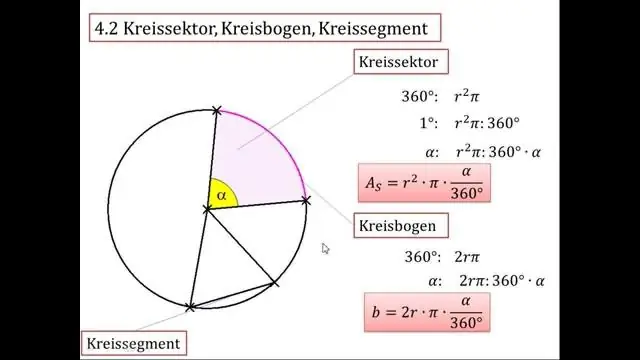
For å beregne radiusen til en sirkel ved å bruke omkretsen, tar du sirkelens omkrets og deler den med 2 ganger π. For en sirkel med en omkrets på 15, deler du 15 med 2 ganger 3,14 og runder av desimaltegnet til svaret ditt på omtrent 2,39
Hvordan kan du identifisere et metall ved hjelp av en flammetest?

Kjemikere bruker det samme prinsippet for å bestemme identiteten til ukjente metaller ved hjelp av en flammetest. Under en flammetest tar kjemikere et ukjent metall og legger det under en flamme. Flammen vil få forskjellige farger basert på hvilket metall som er i stoffet. Forskerne kan deretter identifisere deres ukjente stoff
Hvordan finner du motsatt side av en trekant ved hjelp av Pythagoras?
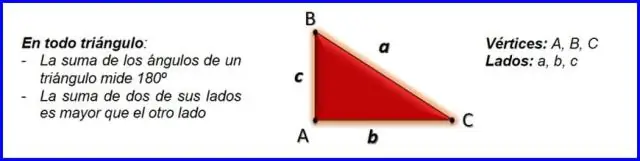
Rette trekanter og Pythagoras teorem Pythagoras teorem, a2+b2=c2, a 2 + b 2 = c 2, kan brukes til å finne lengden på en hvilken som helst side av en rettvinklet trekant. Siden motsatt den rette vinkelen kalles hypotenusen (side c på figuren)
Hvordan skriver du formler ved hjelp av Criss Cross-metoden?
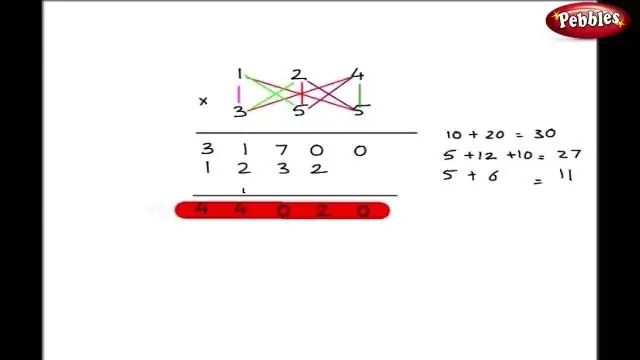
En alternativ måte å skrive en korrekt formel for en ionisk forbindelse på er å bruke kryss-metoden. I denne metoden krysses den numeriske verdien av hver av ioneladningene for å bli subskriptet til det andre ionet. Tegn på anklagene er frafalt. Skriv formelen for bly(IV)oksid
